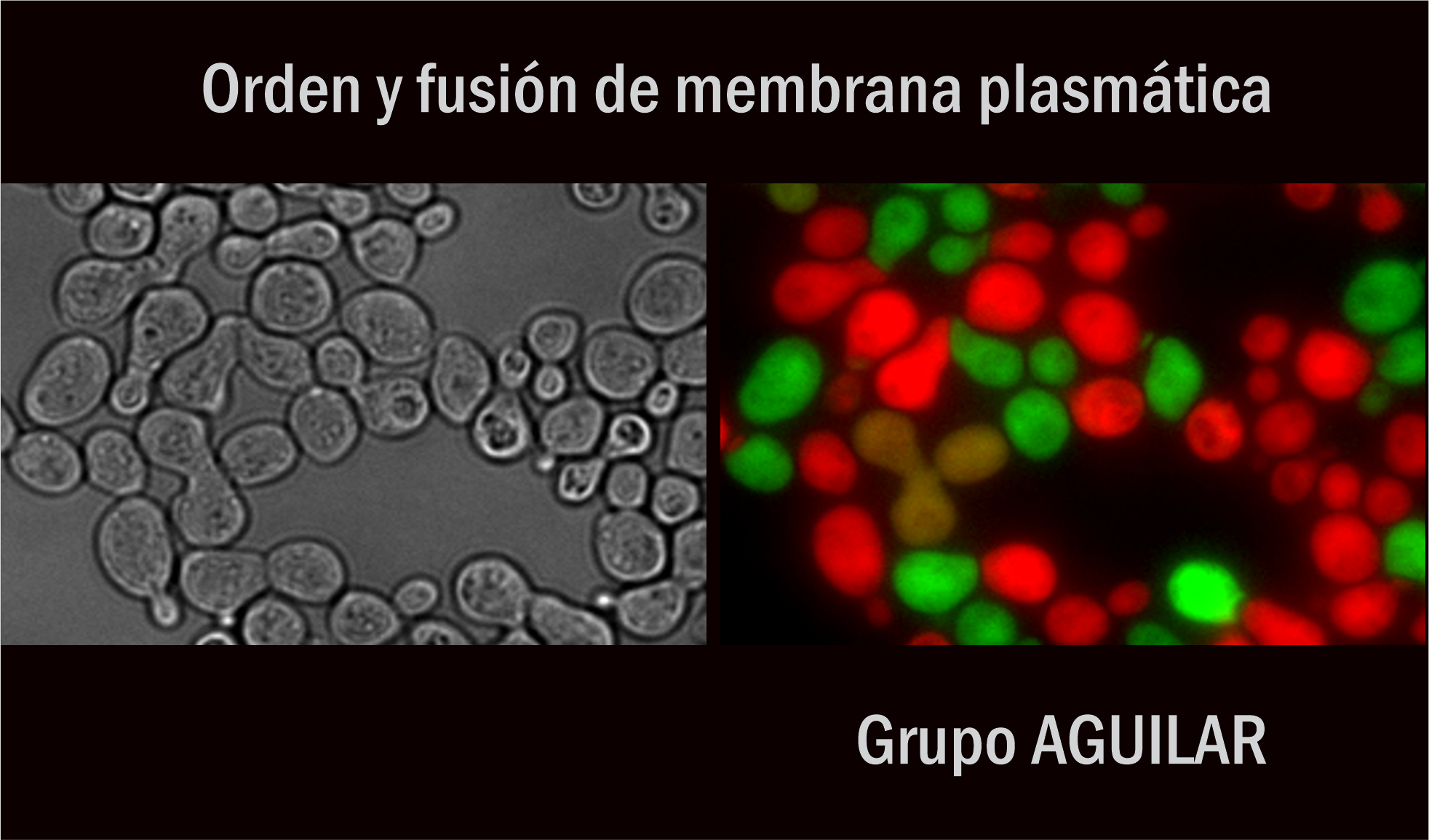
Estudiamos los mecanismos de formación y mantenimiento de nanodominios de membrana plasmática, llamados eisosomas. Nos interesa también determinar cómo estos dominios influyen en la longevidad replicativa de una célula.Por otro lado estudiamos cuales son y cómo actúan las proteínas que catalizan la fusión de las membranas plasmáticas de dos gametos durante la fertilización.
Durante la fertilización dos células sexualmente activas o gametos se fusionan dando lugar a una única célula que integra el material genético de ambas progenitoras.
Identificamos una superfamilia de proteínas, que llamamos fusexinas, que media la fusión de gametos y también la fusión de células somáticas y la entrada de virus a células hospederas1. La familia de fusexinas sexuales parece haberse originado al inicio de la evolución de los eucariotas; están en casi todos los grandes linajes pero no en algunos otros (importantes) como hongos y vertebrados.
Tenemos como objetivos entonces identificar y caracterizar las proteínas que reemplazaron a las fusexinas y fusionan gametos en hongos y vertebrados.
Para eso utilizamos el apareamiento de la levadura Saccharomyces cerevisiae como modelo experimental de fertilización y también distintas herramientas computacionales2 para abarcar todos los eucariotas conocidos.
Por otro lado, encontramos fusexinas en arqueobacterias3. Aún no sabemos que hacen allí pero pensamos que participan de un tipo de sexo primitivo o transferencia horizontal de genes. Para entender la función de fusexinas en arqueobacterias usamos herramientas computacionales y experimentales con arqueas del género Haloferax.
Para poder hacer esto trabajamos en red con grupos de Uruguay, Israel, Suiza y Suecia.
1- Valansi, C., Moi, D.; Leikina, E.; Matveev, E.; Graña, M.; Chernomordik, L; Romero, H.; Aguilar, P.S. and Podbilewicz, B. (2017). Arabidopsis HAP2/GCS1 is a gamete fusion protein homologous to somatic and viral fusogens. J Cell Biol, 216 (3), 571-581, https://doi.org/10.1083/jcb.201610093.
2- Moi, D.; Kilchoer, L.; Aguilar, P.S. and Dessimoz, C. (2020). Scalable Phylogenetic Profiling using MinHash Uncovers Likely Eukaryotic Sexual Reproduction Genes. PLoS Comp Biol., doi: https://doi.org/10.1371/journal.pcbi.1007553.
3- Moi, D.; Nishio, S.; Li, X.; Valansi, C.; Langleib, M.; Brukman, N.; Flyak, K.; Dessimoz, C.; de Sanctis, D.; Tunyasuvunakool, K.; Jumper, J.; Graña, M.; Romero, H.; Aguilar, P.S.; Jovine, L. and Podbilewicz, B. (2022). Discovery of archaeal fusexins homologous to eukaryotic HAP2/GCS1 gamete fusion proteins. Nat Commun, 13 (1): 3880, https://doi.org/10.1038/s41467-022-31564-1.
Estudiamos los eisosomas1,2, nanodominios de membrana plasmática que concentran distintas proteínas transportadoras, de señalización intracelular y otras de función aún desconocida. Los eisosomas participan en la respuesta a diferentes situaciones de estrés (e.g. nutricional, de pared, oxidativo) y en la regulación de la homeostasis de lípidos3. Recientemente encontramos que la pérdida de estos dominios aumenta la longevidad replicativa de S. cerevisiae.
Queremos saber como se forman los eisosomas4, como mantienen su estructura5 y como influyen en la longevidad celular. Para esto utilizamos técnicas de genética molecular y microscopía de fluorescencia acoplada a microfluidica.
En esta línea de trabajo colaboramos con grupos del Departamento de Física de FCEN y de CNEA-Constituyentes.
1- Walther, T.C.; Brickner, J.H.; Aguilar, P.S.; Bernales, S.; Pantoja, C. and Walter, P. (2006) Eisosomes mark static sites of endocytosis. Nature. 439, 998-1003, https://doi.org/10.1038/nature04472.
2- Olivera-Couto, A.; Graña, M.; Harispe, L. and Aguilar, P.S. (2011) The eisosome core is composed of BAR domain proteins. Mol Bio Cell., 22: 2360-2372, https://doi.org/10.1091/mbc.e10-12-1021
3- Aguilar, P.S.; Fröhlich, F.; Rehman, M.; Shales, M.; Ulitsky, I.; Olivera-Couto, A.; Braberg, H.; Shamir, R.; Walter, P.; Mann, M.; Ejsing, C.S.; Krogan, N.J. and Walther, T.C. (2010). A plasma membrane E-MAP Reveals links of the eisosome with sphingolipid metabolism and endosomal trafficking. Nat Struct Mol Biol, 17: 901-908, https://doi.org/10.1038/nsmb.1829.
4- Olivera-Couto, A.; Salzman, V; Mailhos, M.; Digman, M; Gratton, E. and Aguilar, P.S. (2015). Eisosomes are dynamic plasma membrane domains showing Pil1-Lsp1 heteroligomers binding equilibrium. Biophys J, 108: 1633-1644, https://doi.org/10.1016/j.bpj.2015.02.011.
5- Correa Tedesco, F.G.; Aguilar, P.S. and Estrada, L.C. (2022). Correlation analyses reveal differential diffusion behavior of eisosomal proteins between mother and daughter cells. Methods Appl Fluoresc, https://doi.org/10.1088/2050-6120/ac8fe1.
Microscopía confocal y de campo abierto. Videomicroscopía asociada microfluídica. Procesamiento y análisis de imágenes. Diseño y microfabricación de celdas de microfluidica. Herramientas computacionales para detección de homologías lejanas y filogenia molecular.
Técnicas básicas de biología molecular (PCR, RT-PCR, PCR en tiempo real, electroforesis de proteínas y ácidos nucleicos, Western blot, clonado con y sin enzimas), biología celular (inmunofluorescencia, citometría de flujo), bioquímica (transporte y actividad enzimática) y de genética molecular de levaduras y bacterias.
Grupo AGUILAR
“Proyecto chips de microfluídica de alta resolución”
El grupo Aguilar desarrolla el proyecto de “chips de microfluídica de alta resolución” que permiten atrapar y observar mediante videomicroscopía células de levaduras durante toda su vida (aprox. 36h).
Con estas imágenes podemos analizar distintas cepas de interés y estudiar mecanismos que acortan o alargan la longevidad celular.
Integran este proyecto Moises Bustamante @moises.bustamante.505 Becario doctoral – Valentina Salzman, Investigadora Asistente y Pablo Aguilar @luasluba jefe de grupo.
Este es un trabajo conjunto del “Laboratorio de Biología Celular de Membranas” del IFIBYNE dirigido por el Dr. Pablo S. Aguilar, el grupo de la Dra. Laura Estrada del Instituto de Física de Buenos Aires (UBA-CONICET) y el grupo del Dr. Martin Bellino del Instituto de Nanociencia y Nanotecnología (CNEA-CONICET).
Publicaciones (desde 2011)
Datos bibliométricos: ORCID: 0000-0002-9312-2474; Scopus ID: 7005046778 Google Scholar ID: Fx2SUcwAAAAJ&hl
Romero, H.*; Aguilar, P.S.*; Graña, M.; Langelib, M.; Gudiño, V. and Podbilewicz, B. (2024) Membrane fusion and fission during eukaryogenesis. Curr Opin Cell Biol. 86, 102321. https://doi.org/10.
– Moi, D*.; Nishio, S.*; Li, X.*; Valansi, C.; Langleib, M.; Brukman, N.; Flyak, K.; Dessimoz, C.; de Sanctis,D.; Tunyasuvunakool, K.; Jumper, J.; Graña, M.**; Romero, H.**; Aguilar, P.S.**; Jovine, L.** and Podbilewicz, B.**(2022). Discovery of archaeal fusexins homologous to eukaryotic HAP2/GCS1 gamete fusion proteins. Nat Commun, 13 (1): 3880. (highlighted in Science 377, 6606, p 616-617, 2022), https://doi.org/10.1038/s41467-022-31564-1.
– Correa Tedesco, F.G.; Aguilar, P.S. and Estrada, L.C. (2022). Correlation analyses reveal differential diffusion behavior of eisosomal proteins between mother and daughter cells. Methods Appl Fluoresc, https://doi.org/10.1088/2050-6120/ac8fe1.
– Villarruel, C.; Aguilar, P.S. and Ponce Dawson, S. (2021). High rates of calcium-free diffusion in the cytosol of living cells. Biophys J, 120(18):3960-3972, https://doi.org/10.1016/j.bpj.2021.08.019.
– Moi, D.; Kilchoer, L.; Aguilar, P.S. and Dessimoz, C. (2020). Scalable Phylogenetic Profiling using MinHash Uncovers Likely Eukaryotic Sexual Reproduction Genes. PLoS Comp Biol, https://doi.org/10.1371/journal.pcbi.1007553.
– Giorello, F.; Valera, M.J.; Martin, V.; Parada, A.; Salzman, V.; Camesasca, L.; Fariña, L.,; Boido, E.; Medina, K.; Dellacassa, E.; Berna, L.; Aguilar, P.S.; Mas, A.; Gaggero, C. and Carrau, F. (2019). Genomic and Transcriptomic Basis of Hanseniaspora vineae’s Impact on Flavor Diversity and Wine Quality. Appl Environm Microbiol, 85 (1) e01959–18, https://doi.org/10.1128/AEM.01959-18.
– Camesasca, L.; Minteguiaga, M.; Fariña, L.,; Salzman, V.; Aguilar, P.S.; Gaggero, C. and Carrau, F. (2018). Overproduction of isoprenoids by Saccharomyces cerevisiae in a synthetic grape juice medium in the absence of plant genes. Int J Food Microbiol, 282:42- 48, https://doi.org/10.1016/j.ijfoodmicro.2018.05.025.
– Megrian, D., Aguilar, P.S. and Lecumberry, F. (2017). Similarity measure for cell membrane fusion proteins identification. Lecture Notes in Computer Science, 10125, 257-265, https://doi.org/10.1007/978-3-319-52277-7_32.
– Valansi, C*., Moi, D*.; Leikina, E.; Matveev, E.; Graña, M.; Chernomordik, L; Romero, H.; Aguilar, P.S.** and Podbilewicz, B.**. (2017). Arabidopsis HAP2/GCS1 is a gamete fusion protein homologous to somatic and viral fusogens. J Cell Biol, 216 (3), 571-581. * equal contribution, ** corresponding authors, https://doi.org/10.1083/jcb.201610093.
– Carbó, N., Tarkowski, N.; Perez-Ipiña, E., Ponce Dawson, S. and Aguilar, P.S. (2017) Sexual pheromone modulates the frequency of cytosolic Ca2+ bursts in Saccharomyces cerevisiae. Mol Bio Cell. 28(4) 501-510, https://doi.org/10.1091/mbc.E16-07-0481.
– Martin, V.; Giorello, F.; Fariña, L.; Minteguiaga, M.; Salzman, V.; Boido, E.; Aguilar, P.S.; Gaggero, C.; Dellacassa, E.; Mas, A. and Carrau, F. (2016). De Novo Synthesis of Benzenoid Compounds by the Yeast Hanseniaspora vineae Increases the Flavor Diversity of Wines. J Agric Food Chem 64(22):4574-83, https://doi.org/10.1021/acs.jafc.5b05442.
– Bisio, H.; Bonilla, M.; Manta, M.; Graña, M.; Salzman, V.; Aguilar, P.S.; Gladishev, V.N.; Comini, M. and Salinas, G (2015). A new class of thioredoxin-related protein able to bind iron-sulfur clusters. Antiox Redox Signal, https://doi.org/10.1089/ars.2015.6377.
– Salzman, V.; Porro, V.; Bollati-Fogolin, M. and Aguilar, P.S. (2015). Quantitation of Yeast Cell-Cell Fusion Using Multicolor Flow Cytometry. Cytometry A, 87: 843-854; https://doi.org/10.1002/cyto.a.22701.
– Olivera-Couto, A.; Salzman, V; Mailhos, M.; Digman, M; Gratton, E. and Aguilar, P.S. (2015). Eisosomes are dynamic plasma membrane domains showing Pil1-Lsp1 heteroligomers binding equilibrium. Biophys J, 108: 1633-1644, https://doi.org/10.1016/j.bpj.2015.02.011.
– Carrau, F.; Gaggero, C. and Aguilar, P.S. (2015) Yeast diversity and native vigor for flavor phenotypes. Trends Biotechnol 33:148-154, https://doi.org/10.1016/j.tibtech.2014.12.009.
– Trajtenberg, F.; Albanesi, D.; Ruétalo, N.; Botti, H.; Mechaly, A.E.; Nieves, M.; Aguilar, P.S.; Cybulski, L.; Larrieux, N.; de Mendoza, D. and Buschiazzo, A. (2014) Allosteric activation of bacterial response regulators: the role of the cognate histidine kinase beyond phosphorylation. MBio. 18;5(6):e02105, https://doi.org/10.1128/mBio.02105-14.
– Giorello, F.; Berná, L.; Greif, G.; Camesasca, L.; Salzman, V.; Medina, K.; Robello, C.; Gaggero, C.; Aguilar, P.S. and Carrau, F. (2014) Genome sequence of the native apiculate wine yeast Hanseniaspora vineae T02/19AF. Genome Announcements, 2(3). pii: e00530-14, https://doi.org/10.1128/genomeA.00530-14.
– Pizzo, L.; Fariello, M.I.; Lepanto, P.;, Aguilar, P.S. and Kierbel, A. (2014) An image analysis method to quantify CFTR subcellular localization. Mol Cell Probes. pii: S0890-8508(14)00011-5, https://doi.org/10.1016/j.mcp.2014.02.004.
– Zhang, S.; Zheng, H.; Long, N.; Carbó, N.; Chen, P.; Aguilar, P.S. and Lua, L. (2014) FigA, a Putative Homolog of Low-Affinity Calcium System Member Fig1 in Saccharomyces cerevisiae, Is Involved in Growth and Asexual and Sexual Development in Aspergillus nidulans. Eukaryot Cell, 13: 295-303, https://doi.org/10.1128/ec.00257-13.
– Aguilar, P.S.; Baylies, M.K.; Fleissner, A.; Helming, L.; Naozaku, I.; Podbilewicz, B.; Wang, H-M. and Wong, M. (2013) Genetic Basis of cell-cell fusion mechanisms. Trends Genet., 29: 427-437, https://doi.org/10.1016/j.tig.2013.01.011.
– Olivera-Couto, A. and Aguilar, P.S. (2012) Eisosomes and plasma membrane organization. Mol Genet Genomics, 287: 607-620. https://doi.org/10.1007/s00438-012-0706-8.
– Olivera-Couto, A.; Graña, M.; Harispe, L. and Aguilar, P.S. (2011) The eisosome core is composed of BAR domain proteins. Mol Bio Cell., 22: 2360-2372, https://doi.org/10.1091/mbc.e10-12-1021.
Grupos Colaboradores
Laura Estrada, Depto. Física, FCEN-UBA, Argentina.
Silvina Ponce Dawson, Depto. Física, FCEN-UBA, Argentina.
Martin Bellino, CNEA-Centro Atómico Constituyentes, Argentina.
Héctor Romero, Facultad de Ciencias, Universidad de la República, Uruguay.
Martín Graña, Institut Pasteur de Montevideo, Uruguay.
Benjamin Podbilewicz, Technion Institute, Haifa, Israel.
Christophe Dessimoz, David Moi, Université de Lausanne, Suiza.
Luca Jovine, Karolinska Institutet, Suecia.
Mark McCormick, The University of New Mexico, EEUU
dapibus leo.
Integrantes del grupo
Dra. Valentina Salzman
Investigadora Asistente
Dra. Valentina Salzman
Investigadora Asistente
Publicaciones
2020 Genetic structure of Photosystem II functionality in rice unraveled by GWAS analysis. BioRxiv 2020.09.28.317479. Vilas JM¥, Burgos E¥, Puig ML, Colazo J, Livore A, Ruiz AO, Carrari F, Rodriguez AA, Maiale SJ. DOI: https://doi.org/10.1101/2020.09.28.317479.
2019 Fruit metabolic and transcriptional programs differentiate among Andean tomato Solanum lycopersicum L.) accessions. Planta, PLAA-D-19-00502. D´Angelo M¥, Zanor MI¥, Burgos E, Asprelli P, Boggio SB, Carrari F, Peralta IE,4, Valle E. DOI: https://doi.org/10.1007/s00425-019-03274-4.
2018 Pre-breeding studies in Panicum coloratum var. coloratum: Characterization using agro-morphological traits and molecular markers. Burgos E, Thompson C, Giordano M, Tomás MA. Tropical Grasslands-Forrajes Tropicales. 2018; 6(2):82–92.DOI: https://doi.org/10.17138/tgft(6)82-92.
Research Interests Much of my current research focuses on bioinformatic analysis particularly for Genome-Wide Association Studies, QTL mapping and RNA-seq data. The main goal of my project is to understand the regulation of tococromanols (vitamin E) compound-derived metabolic pathways. Part of this goal involve the study one of the last steps in tococromanols biosynthesis which is catalyzed by the 2-methyl-6-phytylquinol methyltransferase. This enzyme in tomato is encoded by two key paralog genes, VTE3(1) and VTE3(2). I intend to integrate different types of high-throughput data analysis to elucidate how tomato plants, as sessile organisms, had developed a plethora of response mechanisms as tococromanols to environmental constraints. The regulation of those mechanisms can be explained at different organization levels; from changes in leaf anatomy to epigenomics.
Publications
2020 Genetic structure of Photosystem II functionality in rice unraveled by GWAS analysis. BioRxiv 2020.09.28.317479. Vilas JM¥, Burgos E¥, Puig ML, Colazo J, Livore A, Ruiz AO, Carrari F, Rodriguez AA, Maiale SJ. DOI: https://doi.org/10.1101/2020.09.28.317479.
2019 Fruit metabolic and transcriptional programs differentiate among Andean tomato Solanum lycopersicum L.) accessions. Planta, PLAA-D-19-00502. D´Angelo M¥, Zanor MI¥, Burgos E, Asprelli P, Boggio SB, Carrari F, Peralta IE,4, Valle E. DOI: https://doi.org/10.1007/s00425-019-03274-4.
2018 Pre-breeding studies in Panicum coloratum var. coloratum: Characterization using agro-morphological traits and molecular markers. Burgos E, Thompson C, Giordano M, Tomás MA. Tropical Grasslands-Forrajes Tropicales. 2018; 6(2):82–92.DOI: https://doi.org/10.17138/tgft(6)82-92.
-- Estanislao Burgos, Postdoctoral researcher IFIBYNE-UBA-CONICET Instituto de Fisiología Biología Molecular y Neurociencias Ciudad Universitaria - Pabellón IFIBYNE (C1428EHA) Buenos Aires - Argentina Tel: 54-11-4576-3386 /4576-3368 E-MAIL: eburgos@fbmc.fcen.uba.ar ; burgosestanislao@gmail.com
- Phone:+549 (11) 2546-6589
- Email:info@example.com
Virginia Gudiño
Becaria Doctoral
Virginia Gudiño
Becaria Doctoral
- Phone:+549 (11) 2546-6589
- Email:info@example.com
Moises Bustamante
Becario Doctoral
Moises Bustamante
Becario Doctoral
- Phone:+549 (11) 2546-6589
- Email:info@example.com
Nahuel Tarkowski
Cargo del Investigador
Nahuel Tarkowski
Cargo del Investigador
- Phone:+549 (11) 2546-6589
- Email:info@example.com
Miembros Anteriores
Lic. Francisco Correa Tedesco, Microfabrication Specialist Stämm
Dr. David Moi, posdoc Université de Lausanne, Suiza
Dra. Cecilia Villarruel, Clinical Research Associate, Syneos Health
Dr. Juan Eizaguirre, Consultor Cervecero Eukarya
Dr. Federico Perez de Berti, CPA IMBICE-CONICET
Dra. Daniela Megrian, posdoc Institut Pasteur Paris
Dra. Natalia Carbo
MSc. Milagros Mailhos
MSc. Marcos Nieves, estudiante doctorado Universität Münster, Alemania
Dra. Agustina Olivera-Couto, Oceánica-Levtec, Uruguay
Lic. Micaela Nievas, LIAN-FLENI