Nuestro laboratorio intenta comprender los mecanismos moleculares a través de los cuales los factores de transcripción regulan la expresión génica. Usando técnicas avanzadas de microscopía de fluorescencia y el receptor de glucocorticoides como modelo de estudio, encontramos que el ADN puede inducir un cambio conformacional en los factores de transcripción, alterando su estructura cuaternaria. También estudiamos cómo el movimiento y localización de los factores de transcripción dentro del núcleo celular afecta la actividad transcripcional de los mismos.
Líneas de Investigación:
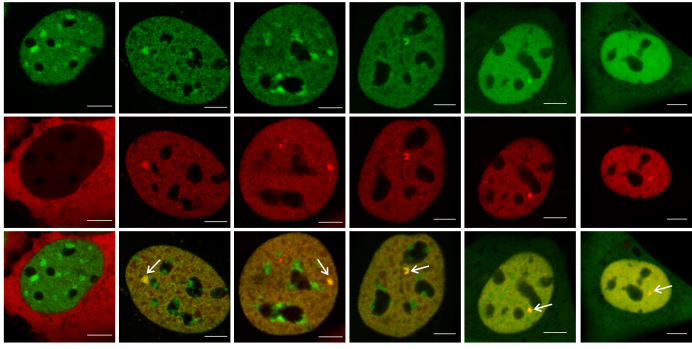
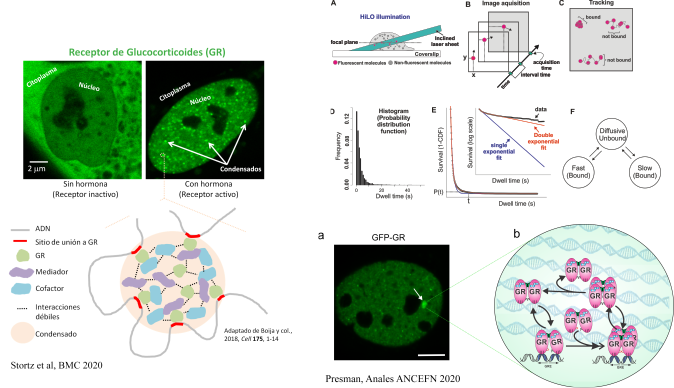
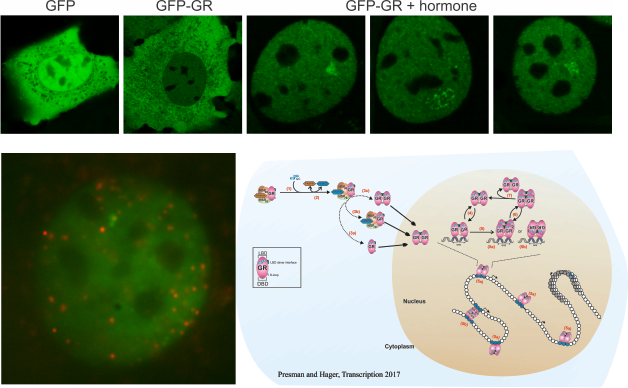
El receptor de glucocorticoides (GR), un factor de transcripción activado por ligando, constituye uno de los principales blancos moleculares para el desarrollo de fármacos debido a su potente efecto antiinflamatorio e inmunosupresor. Desafortunadamente, si bien varias enfermedades son tratadas mediante el uso de estas drogas, ninguna de ellas puede considerarse libre de graves efectos secundarios. Es por ello que durante las últimas décadas se han realizado enormes esfuerzos por encontrar ligandos disociados, es decir, hormonas sintéticas que separen los efectos deseados (antiinflamatorios) de los efectos adversos (metabólicos). El paradigma vigente establece una relación directa entre el estado de oligomerización del GR (si actúa como dímero o como monómero) y su actividad fisiológica (efectos antiinflamatorios o metabólicos). Este modelo disociado ha dirigido la búsqueda de ligandos “más seguros” durante los últimos 20 años, sin mayores éxitos. Desde hace algunos años, hemos venido desafiando el modelo vigente al sugerir que la dicotomía dímeromonómero es inexistente in vivo, y que el GR sufre una transición dímero->tetrámero, propuesta como forma activa final del receptor. Nuestro laboratorio tiene como objetivo dilucidar la relación funcional entre la estructura cuaternaria del GR, su dinámica intranuclear, y su actividad transcripcional.
Papers destacados en esta línea de trabajo:
– The mineralocorticoid receptor forms higher order oligomers upon DNA binding. Gregory Fettweis, Thomas A. Johnson, Brian Almeida-Prieto, Julián Weller-Pérez, Diego M. Presman, Gordon L. Hager✉, Diego Alvarez de la Rosa✉. Protein Sci. 2024 33(3):e4890. https://doi.org/10.1002/pro.4890
– The multivalency of the glucocorticoid receptor ligand-binding domain explains its manifold physiological activities. Alba Jiménez-Panizo#, Andrea Alegre-Martí#, Theophilus Tettey#, Gregory Fettweis, Montserrat Abella, Rosa Antón, Thomas A Johnson, Sohyoung Kim, R. Louis Schiltz, Israel Nuñez-Barrios, Joan Font-Díaz, Carme Caelles, Annabel F. Valledor, Paloma Pérez, Ana M. Rojas, Juan Fernández-Recio, Diego M. Presman, Gordon L. Hager✉, Pablo Fuentes-Prior✉, and Eva Estébanez-Perpiñá✉. #co-first authors. ✉corresponding authors. Nucleic Acids Research, 2022; (50) 22:13063-13082. https://doi.org/10.1093/nar/gkac1119.
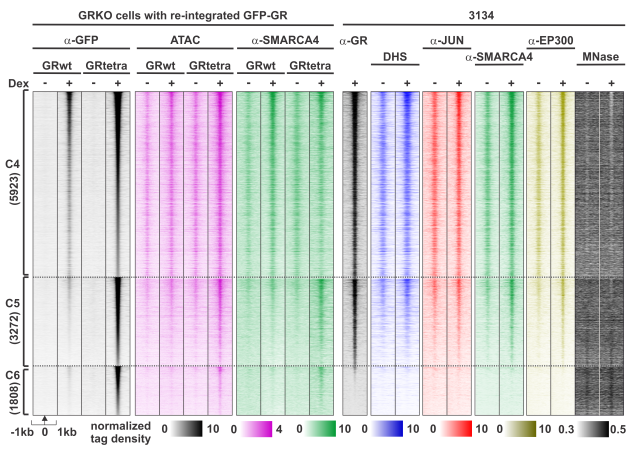
– Genome-wide Binding Potential and Regulatory Activity of the Glucocorticoid Receptor’s Monomeric and Dimeric Forms. Thomas A. Johnson; Ville Paakinaho, Sohyoung Kim, Gordon L. Hager, and Diego M. Presman. Nature Communications 2021, 12, 1987. https://doi.org/10.1038/s41467-021-22234-9.
– Glucocorticoid Receptor Quaternary Structure Drives Chromatin Occupancy and Transcriptional Outcome. Ville Paakinaho, Thomas A. Johnson, Diego M. Presman, Gordon L. Hager. Genome Research, 2019 Aug;29(8):1223-1234. https://doi.org/10.1101/gr.244814.118.
– DNA-binding Triggers Tetramerization of the Glucocorticoid Receptor in Live Cells. Presman, D.M, Ganguly, S., Schiltz, R.L., Johnson, T.A., Karpova, T.S., Hager, G.L. Proc Natl Acad Sci USA, 2016 113 (29) 8236-8241. co-corresponding authors. https://doi.org/10.1073/pnas.1606774113.
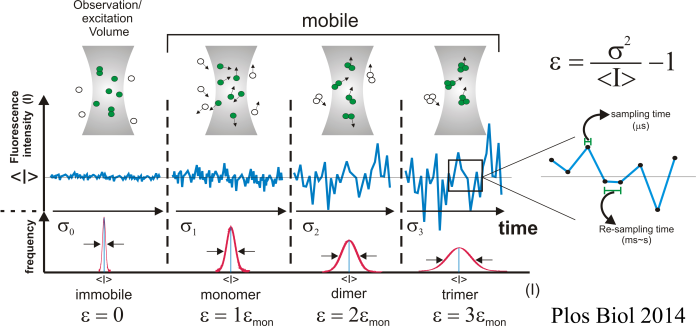
– Live cell imaging unveils multiple domain requirements for in vivo dimerization of the glucocorticoid receptor. Presman, D.M., Ogara, M.F, Stortz, M., Alvarez, L.D., Pooley, J.R., Schiltz, R.L, Grøntved, L., Johnson, T.A., Mittelstadt, P.R., Ashwell, J.D., Ganesan, S., Burton, G., Levi, V., Hager, G.L., Pecci, A. PLoS Biology 2014 ;12(3):e1001813. Destacada en F1000. https://doi.org/10.1371/journal.pbio.1001813.
Durante décadas, se creyó que los factores de transcripción interactuaban con sus sitios blanco de manera estática, funcionalmente activos por minutos o incluso horas. Sin embargo, este paradigma fue cuestionado cuando pudo observarse la dinámica de estas proteínas mediante microscopía de fluorescencia en células vivas. Mas recientemente, los grandes avances tecnológicos en materia de microscopía cuantitativa están permitiendo la visualización directa de moléculas individuales con resoluciones temporales y espaciales sin precedentes, permitiendo caracterizar los patrones de búsqueda y unión de los factores de transcripción a sus sitios blanco. Uno de los objetivos de nuestro laboratorio es dilucidar la relación que existe entre la dinámica de los factores de transcripción a nivel de moléculas individuales con su actividad transcripcional. Utilizamos los receptores de esteroides como modelo biológico debido a que su actividad es ligando-dependiente y poseen enorme relevancia fisiológica y farmacológica.
Papers destacados en esta línea de trabajo:
– Chromatin structure and dynamics: one nucleosome at a time. Diego M. Presman✉, Belén Benitez, Agustina Lafuente, Alejo Vazquez Lareu. Histochem Cell Biol (2024). https://doi.org/10.1007/s00418-024-02281-1.
–Dynamic switching of transcriptional regulators between two distinct low-mobility chromatin states. Kaustubh Wagh†, Diana A Stavreva†, Rikke AM Jensen, Ville Paakinaho, Gregory Fettweis, R Louis Schiltz, Daniel Wüstner, Susanne Mandrup, Diego M Presman✉, Arpita Upadhyaya✉, Gordon L Hager✉. Science Advances 2023, 9:eade1122. †co-first authors. ✉corresponding authors. https://www.science.org/doi/10.1126/sciadv.ade1122
– An intrinsically disordered region-mediated confinement state contributes to the dynamics and function of transcription factors. David A. Garcia, Thomas A. Johnson*, Diego M. Presman*, Gregory Fettweis, Kaustubh Wagh, Lorenzo Rinaldi, Diana A. Stavreva, Ville Paakinaho, Rikke A.M. Jensen, Susanne Mandrup, Arpita Upadhyaya, and Gordon L. Hager. Molecular Cell, 2021, 81:1484-1498.
– Power-law behaviour of transcription factor dynamics at the single-molecule level implies a continuum affinity model. David A. Garcia*, Gregory Fettweis*, Diego M. Presman*, Ville Paakinaho, Christopher Jarzynski, Arpita Upadhyaya, and Gordon L. Hager. Nucleic Acids Research, 2021, Feb17, Online ahead of print. *co-first authors.. https://doi.org/10.1093/nar/gkab072.
– Transcriptional Bursting and Co-bursting Regulation by Steroid Hormone Release Pattern and Transcription Factor Mobility. Diana A. Stavreva, David A. Garcia, Gregory Fettweis, Prabhakar R. Gudla, George F. Zaki, Vikas Soni, Andrew McGowan, Geneva Williams, Anh Huynh, Murali Palangat, R. Louis Schiltz, Thomas A. Johnson, Diego M. Presman, Matthew L. Ferguson, Gianluca Pegoraro, Arpita Upadhyaya, and Gordon L. Hager. Molecular Cell, 2019 Jul 26. pii: S1097-2765(19)30499-X. https://doi.org/10.1016/j.molcel.2019.06.042.
– Steroid Receptors Reprogram FoxA1 Occupancy through Dynamic Chromatin Transitions. Swinstead, E.E., Miranda, T.B., Paakinaho, V., Baek, S., Goldstein, I., Hawkins, M., Karpova, T.S., Ball, D., Mazza, D., Lavis, L.D., Grimm, J.B., Morisaki, M., Grøntved, L., Presman, D.M., and Hager, G.L. Cell, 2016, 165:593-605. Destacada en F1000. https://doi.org/10.1016/j.cell.2016.02.067
El núcleo celular contiene diversos compartimientos funcionales que no se encuentran delimitados por membranas. Su valor adaptativo radicaría en permitir tanto la modulación de la cinética como la especificidad de numerosas reacciones bioquímicas que ocurren dentro del núcleo, incluyendo el proceso de transcripción. Recientemente, se ha propuesto que muchos de estos compartimientos se formarían como consecuencia de un proceso de separación de fases líquido-líquido. Este nuevo paradigma inició una reevaluación de la compleja distribución de numerosas biomoléculas involucradas en la transcripción. Nuestro objetivo es comprender el rol funcional y regulatorio de los condensados intranucleares en el proceso de transcripción, utilizando al GR como modelo de estudio. Mediante la generación de herramientas moleculares basadas en las tecnologías de CRISPR y optogenética, y a través del uso de técnicas avanzadas de microscopía de fluorescencia, estamos evaluando el proceso de formación de los condensados de GR y su rol en la modulación de la transcripción.
Papers destacados en esta línea de trabajo
– Transcriptional Condensates: A Blessing or a Curse for Gene Regulation? Martin Stortz, Diego M. Presman✉, and Valeria Levi✉. Communications Biology 2024, 7:187. https://doi.org/10.1038/s42003-024-05892-5
– SOX2 modulates the nuclear organization and transcriptional activity of the glucocorticoid receptor. Martin Stortz, Camila Oses, Camila Vázquez Echegaray, Adali Pecci, Alejandra Guberman, Diego M. Presman✉ and Valeria Levi✉. Journal of Molecular Biology 2022 Oct 26;167869. Online ahead of print. ✉corresponding authors. https://doi.org/10.1016/j.jmb.2022.167869.
– Phasing the intranuclear organization of steroid hormone receptors. Martin Stortz*




–Unraveling the Molecular Interactions Involved in Phase Separation of Glucocorticoid Receptor. Martin Stortz, Adali Pecci, Diego Presman


–Mapping the Dynamics of the Glucocorticoid Receptor within the Nuclear Landscape. Stortz M, Presman DM, Bruno L, Annibale P, Dansey MV, Burton G, Gratton E, Pecci A, Levi V. Sci Rep. 2017, 7(1):6219. https://doi.org/10.1038/s41598-017-06676-0.
Principalmente usamos técnicas avanzadas de microscopía de fluorescencia, incluyendo la técnica de Número y Brillo. También utilizamos técnicas básicas de biología molecular (clonado, PCR, RT-PCR, PCR en tiempo real, Western), cultivo de células de mamíferos, transfecciones transitorias. Por último, hacemos experimentos de genómica (ChIP-seq, RNA-seq, ATAC-seq) en colaboración con el grupo del Dr. Hager (NIH)
– Chromatin structure and dynamics: one nucleosome at a time. Diego M. Presman✉, Belén Benitez, Agustina Lafuente, Alejo Vazquez Lareu. Histochem Cell Biol (2024). https://doi.org/10.1007/s00418-024-02281-1
– The mineralocorticoid receptor forms higher order oligomers upon DNA binding. Gregory Fettweis, Thomas A. Johnson, Brian Almeida-Prieto, Julián Weller-Pérez, Diego M. Presman, Gordon L. Hager✉, Diego Alvarez de la Rosa✉. Protein Sci. 2024 33(3):e4890. https://doi.org/10.1002/pro.4890
– Transcriptional Condensates: A Blessing or a Curse for Gene Regulation? Martin Stortz, Diego M. Presman✉, and Valeria Levi✉. Communications Biology 2024, 7:187. https://doi.org/10.1038/s42003-024-05892-5
– Dynamic switching of transcriptional regulators between two distinct low-mobility chromatin states. Kaustubh Wagh†, Diana A Stavreva†, Rikke AM Jensen, Ville Paakinaho, Gregory Fettweis, R Louis Schiltz, Daniel Wüstner, Susanne Mandrup, Diego M Presman✉, Arpita Upadhyaya✉, Gordon L Hager✉. Science Advances 2023, 9:eade1122. †co-first authors. ✉corresponding authors. https://www.science.org/doi/10.1126/sciadv.ade1122
– SOX2 modulates the nuclear organization and transcriptional activity of the glucocorticoid receptor#. Martin Stortz, Camila Oses, Camila Vázquez Echegaray, Adali Pecci, Alejandra Guberman, Diego M. Presman✉ and Valeria Levi✉. Journal of Molecular Biology 2022; 434(24):167869. ✉corresponding authors. https://doi.org/10.1016/j.jmb.2022.167869. #Tapa de la revista
– The multivalency of the glucocorticoid receptor ligand-binding domain explains its manifold physiological activities. Alba Jiménez-Panizo#, Andrea Alegre-Martí#, Theophilus Tettey#, Gregory Fettweis, Montserrat Abella, Rosa Antón, Thomas A Johnson, Sohyoung Kim, R. Louis Schiltz, Israel Nuñez-Barrios, Joan Font-Díaz, Carme Caelles, Annabel F. Valledor, Paloma Pérez, Ana M. Rojas, Juan Fernández-Recio, Diego M. Presman, Gordon L. Hager✉, Pablo Fuentes-Prior✉, and Eva Estébanez-Perpiñá✉. #co-first authors. ✉corresponding authors. Nucleic Acids Research, 2022, (50) 22:13063-13082. https://doi.org/10.1093/nar/gkac1119.
– Genome-wide Binding Potential and Regulatory Activity of the Glucocorticoid Receptor’s Monomeric and Dimeric Forms. Thomas A. Johnson; Ville Paakinaho, Sohyoung Kim, Gordon L. Hager


 , Diego M. Presman*
, Diego M. Presman* , Adali Pecci
, Adali Pecci , and Valeria Levi
, and Valeria Levi . Biochemical Journal 2021, 478(2):443-461. https://doi.org/10.1042/BCJ20200883 *Co-first authors;
. Biochemical Journal 2021, 478(2):443-461. https://doi.org/10.1042/BCJ20200883 *Co-first authors;  corresponding authors.
corresponding authors. , and Valeria Levi
, and Valeria Levi . BMC Biology, 2020, 18(1):59. https://doi.org/10.1186/s12915-020-00788-2.
. BMC Biology, 2020, 18(1):59. https://doi.org/10.1186/s12915-020-00788-2.  co-corresponding authors
co-corresponding authors , Gordon L. Hager
, Gordon L. Hager . Genome Research, 2019 Aug;29(8):1223-1234. https://doi.org/10.1101/gr.244814.118.
. Genome Research, 2019 Aug;29(8):1223-1234. https://doi.org/10.1101/gr.244814.118.  co-corresponding authors. Destacada en F1000.
co-corresponding authors. Destacada en F1000. ., Hager, G.L. Transcription, 2017. 8:1, 32-39, https://doi.org/10.1080/21541264.2016.1249045.
., Hager, G.L. Transcription, 2017. 8:1, 32-39, https://doi.org/10.1080/21541264.2016.1249045.  corresponding author.
corresponding author. , Ganguly, S., Schiltz, R.L., Johnson, T.A., Karpova, T.S., Hager, G.L
, Ganguly, S., Schiltz, R.L., Johnson, T.A., Karpova, T.S., Hager, G.L . Proc Natl Acad Sci USA, 2016 113 (29) 8236-8241.
. Proc Natl Acad Sci USA, 2016 113 (29) 8236-8241.  co-corresponding authors. https://doi.org/10.1073/pnas.1606774113
co-corresponding authors. https://doi.org/10.1073/pnas.1606774113Integrantes del grupo

Lic. Agustina LaFuente (Becaria Doctoral CONICET). Oligomerización y actividad del GR

Lic. Belén Benitez Dos Santos (Becaria Doctoral CONICET). Bio-condensados del GR

Alejo Vázquez Lareu (Est. Licenciatura). Interacción de proteínas por FRET
Miembros Anteriores
1. Sebastián Yanichevsky (Tesis de licenciatura)
2. Stephanie Junge (Tesis de Licenciatura)